|
|
|||||
Accueil > Actualités > L'Actu des membres et partenaires > GENOPOLE
Juillet 2019 - La biologie à l'ère du numérique grâce à un calculateur de 4 Tflops ouvert à la communauté scientifique et industrielle du biocluster Genopole L'Atelier de Biologie de synthèse [abSYNTH] est hébergé au plus près de l'Unité de Génomique Métabolique (UMR 8030 CEA / CNRS / UEVE) et en particulier du Laboratoire de Biologie systémique et synthétique (LSSB). L'abSYNTH a pour objectif de proposer de façon modulaire l'ensemble des tâches d'un projet de biologie de synthèse, de la conception jusqu'à sa réalisation. La singularité de cette plate-forme Genopole repose sur l'intégration en un lieu unique de l'ensemble des outils nécessaires au développement d'un projet de biologie de synthèse. Trois modules constitutifs forment l'ossature de la plate-forme. Ces modules correspondent aux phases de conception, de construction et de caractérisation de nouveaux circuits génétiques. Ils s'appliquent à plusieurs échelles, depuis les bio-briques moléculaires jusqu'aux dispositifs et systèmes régulatoires et métaboliques. Concernant le premier module, conception, l'abSYNTH dispose d'un calculateur configuré pour répondre aux besoins des biologistes qui peut développer une puissance en crête de 4 TFlops (4 000 milliards d'opérations par seconde) et récemment, sa capacité de stockage a été multipliée par 15 pour l'amener jusqu'à 30 To. Financé conjointement par le Conseil départemental de l'Essonne, Genopole et la Région Ile-de-France, ce calculateur est ouvert à l'ensemble de la communauté scientifique et industrielle du biocluster Genopole. L'Atelier de biologie de synthèse dispose d’équipements nécessaires à la conception, la construction et la caractérisation de systèmes biologiques complexes basés sur, ou inspirés par le vivant mais dotés de fonctions absentes dans la nature et d'autre part, la conception automatique de circuits génétiques. CONCEPTION : offre logicielle et outil de calcul intensif pour la conception de biobriques moléculaires et de dispositifs et systèmes régulatoires et métaboliques. Aujourd'hui, 10 algorithmes ou logiciels sont accessibles sur absynth.issb.genopole.fr. CONSTRUCTION : chaîne d'assemblage automatisée de biobriques par des méthodes de biologie moléculaire permettant la production de clones d’ADN : clonage à haut-débit en phase liquide ; robot pipeteur ; appareils PCR et tous les équipements de laboratoires nécessaires à cette activité. CARACTERISATION : Procédé partiellement automatisé de tests phénotypiques des assemblages réalisés dans des microorganismes comme la bactérie ou la levure, en utilisant un spectromètre de masse (Q-TOF), un appareil de PCR quantitative, des systèmes de documentation de gel, une électrophorèse capillaire sur nanopuce, plusieurs lecteurs multimodaux de plaques (96-, 384- et 1536 puits), etc. Pour obtenir des informations, contacter Julien.Picot@genopole.fr |
|
| Juin 2019 -
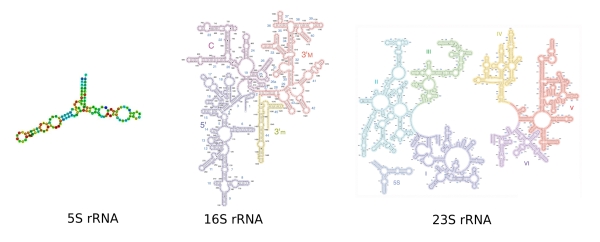
Le biocluster Genopole dispose d'une plate-forme logicielle dédiée à l'étude des ARN non-codants dont l'importance est aujourd'hui bien connue tant par leur nombre, leur diversité et leur rôle dans le fonctionnement cellulaire. En effet, les ARN non-codants, issus de la transcription de l’ADN et non traduits en protéine par les ribosomes, sont des régulateurs du contrôle de l'expression génique, de l'établissement des domaines chromatiniens et de la stabilité du génome. Ils interviennent dans divers processus biologiques et certains d'entre eux, notamment les microARN, sont connus pour être impliqués dans de nombreuses maladies telles que le cancer et les maladies neurodégénératives. Leur étude permet de mieux comprendre ces pathologies mais aussi d'envisager de nouvelles approches thérapeutiques pour les maladies génétiques et le cancer. La plate-forme EvryRNA (https://EvryRNA.ibisc.univ-evry.fr), soutenue par Genopole, est un serveur web mettant à disposition de la communauté scientifique différents logiciels bio-informatiques de prédiction de structure secondaire d'ARN, d'identification et de recherche à grande échelle dans des séquences génomiques d'ARN non-codants, notamment les petits ARN (microARN, piARN, etc.). Tous les logiciels disponibles sur la plate-forme EvryRNA ont fait l’objet d’articles publiés dans des revues scientifiques internationales à fort impact factor telles que Nucleic Acides Research, Bioinformatics, RNA, BMC Bioinformatics et d’autres. Voici une brève description des outils bio-informatiques :
Ces logiciels, tous développés au sein du laboratoire "Informatique, Biologie Intégrative et Systèmes Complexes" [IBISC] de l'Université d'Evry Paris-Saclay, correspondent à différents algorithmes basés sur des approches d'appariement de modèles et d'algorithmes de séquence, ainsi que des approches d'apprentissage automatique. Fariza TAHI, chercheur au laboratoire IBISC et responsable de la Plate-forme EvryRNA indique que "comparés aux logiciels concurrents, les outils de la plate-forme EvryRNA apportent, en plus de l'efficacité des prédictions, la rapidité nécessaire à des analyses à grande échelle". Pour obtenir plus d'information sur les logiciels disponibles, veuillez contacter Fariza.Tahi@ibisc.univ-evry.fr |
|
| 4 septembre 2015 -
La plate-forme EvryRNA de Genopole est un serveur web mettant à disposition de la communauté scientifique les différents algorithmes et outils bioinformatiques développés au sein du laboratoire « Informatique, Biologie Intégrative et Systèmes Complexes » (www.ibisc.univ-evry.fr) de l’Université d’Evry-Val-d’Essonne, dédiés à de l’analyse génomique, et plus précisément à la prédiction et l’analyse des ARNs (acides ribonucléiques) non-codants. Les ARNs non-codants sont des molécules issues de la transcription de l’ADN qui ne seront pas traduites en protéine par les ribosomes. Ces ARNs sont des régulateurs du contrôle de l’expression des gènes, de l’établissement des domaines chromatiniens et de la stabilité du génome. Ils interviennent dans différents processus biologiques, et certains d’entre eux, notamment les microARNs, sont connus pour être impliqués dans un grand nombre de maladies, telles que le cancer et les maladies neuro-dégénératives. Leur étude permet de mieux comprendre le fonctionnement du vivant, notamment la différenciation et la prolifération cellulaire, mais aussi d'envisager de nouvelles approches thérapeutiques pour les maladies génétiques et le cancer. La plate-forme logicielle EvryRNA regroupe de nombreux logiciels bioinformatiques de prédiction de structure secondaire d'ARNs, d'identification et de prédiction d'ARNs non-codants notamment les petits ARNs (microARNs, piARNs, etc) à grande échelle dans des séquences génomiques :
Ces logiciels, correspondants à différents algorithmes utilisant des approches d’algorithmique de séquences et d’apprentissage automatique, sont tous publiés dans des revues scientifiques internationales à fort impact facteur (Nucleic Acids Research, Bioinformatics, RNA, …). Ils ont comme particularité par rapport aux logiciels concurrents la rapidité, permettant ainsi des analyses à grande échelle, en plus de l’efficacité des prédictions. Par ailleurs, on peut trouver sur la plate-forme EvryRNA d’autres outils développés en annexe, tels que :
Pour obtenir des informations complémentaires sur la plate-forme EvryRNA (http://EvryRNA.ibisc.univ-evry.fr/), veuillez contacter Madame Fariza TAHI via fariza.tahi@ibisc.univ-evry.fr
|
|
2 septembre 2015 - Situé au cœur du biocluster Genopole (Evry - 91), l'Institut de Biologie Systémique et Synthétique (iSSB) dispose d'une plate-forme collaborative et de service unique nommée Atelier de biologie de synthèse [abSYNTH]. Créé en 2010, l'iSSB (CNRS FRE3561, Université d’Évry-Val-d'Essonne, Genopole) est le premier laboratoire français dédié à la Biologie de Synthèse. La Biologie de Synthèse est la construction et la caractérisation de systèmes biologiques complexes basés sur ou inspirés par le vivant mais dotés de fonctions absentes dans la nature. Concernant l’étape de conception, phase amont de la construction, l'abSYNTH met à disposition une bibliothèque de logiciels (absynth.issb.genopole.fr) développés au sein de l’iSSB dont certains exploitant un calculateur (4 TFlops). Ce calculateur devrait à terme intégrer le mésocentre de calcul du bassin évryen (environ 20 TFlops) financé par la Région Ile-de-France et qui verra le jour en 2016 !
|
|